こちらにおいて、
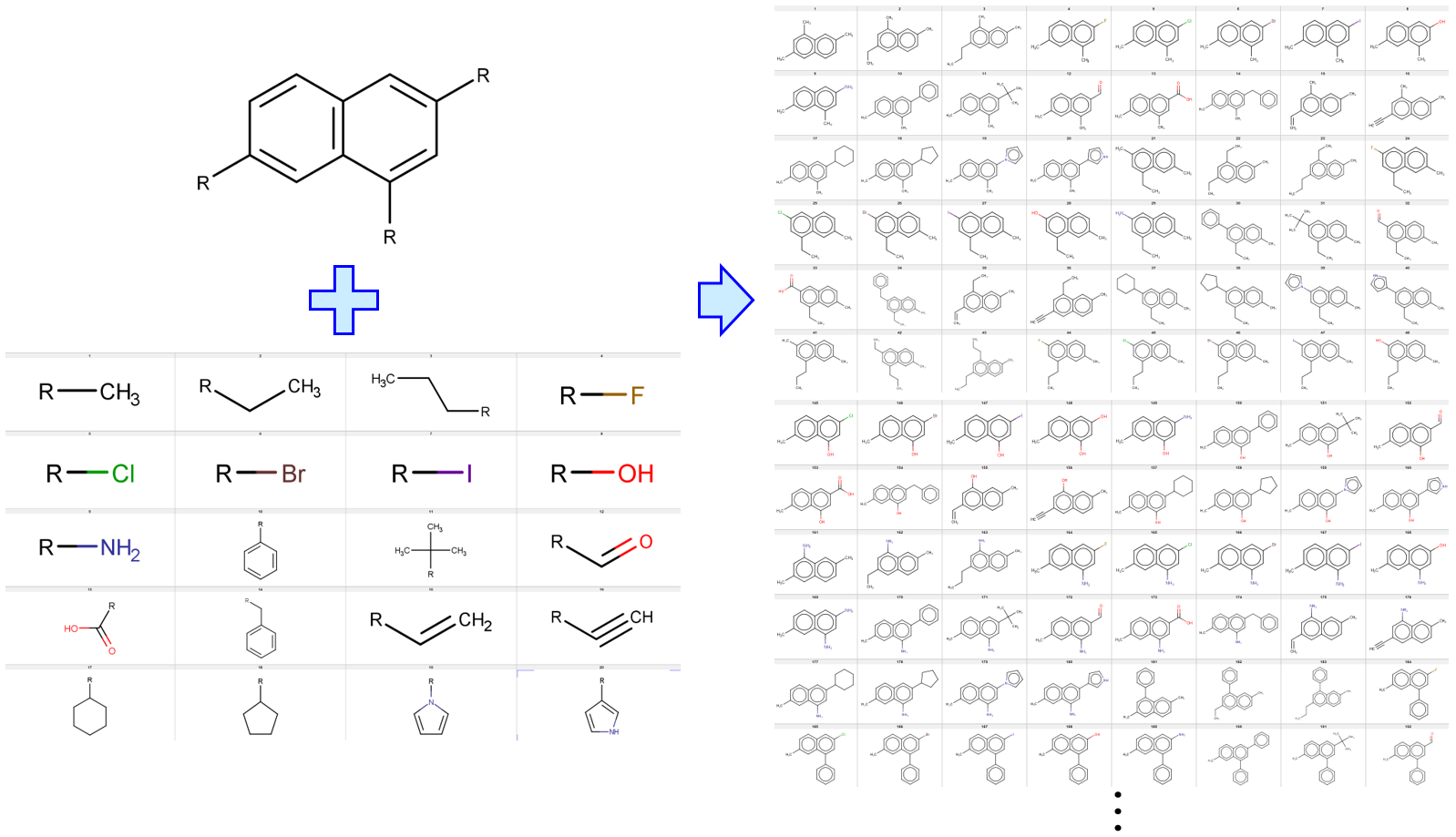
誘導体の化学構造を自動生成するプログラム Structure Generator based on R-Group (SGRG) の話をしました。
今回は SGRG を活用してどんなことができるのか、について書いておきます。
1. 主骨格はそのままで、側鎖を変えた化学構造を生成できる
これはそのままですね。事前に定量的構造活性相関 (Quantitative Structure Activity Relationship, QSAR) モデルや、定量的構造物性相関 (Quantitative Structure Property Relationship, QSPR) モデルを構築しておけば、生成させた化学構造を QSAR・QSPR モデルに入力することで、それらの化学構造がもつ活性値や物性値を推定できます。推定結果に基づいて、高い活性をもつと考えられる化学構造など、次に合成する化学構造を選ぶわけです。
もちろん、主骨格を変えれば別の化学構造を生成できますので、いろいろな化学構造を作れます。
2. QSAR・QSPR モデルを解釈できる
化学構造と記述子との間の関係が非線形だったり、記述子と活性・物性との間の関係が非線形だったりして、QSAR モデルや QSPR モデルを解釈することはなかなか難しいです。
SGRG を用いることで、主骨格はそのままで、側鎖だけを変えた化学構造を生成できます。これらの化学構造をすべて QSAR モデルや QSPR モデルに入力して、出力された活性値や物性値を確認することで、側鎖の活性や物性に対する影響を見ることができます。つまり QSAR モデルや QSPR モデルはブラックボックスでも、化学構造の一部を変えたものの推定結果を比較して、モデルを解釈するわけです。
側鎖を変えて合成して、ということを繰り返して側鎖の影響を調べることを、実際に実験するのではなくモデルで行っている感じです。
注意点は2つあります。
1 つ目として、モデルの適用範囲 (Applicability Domain, AD)
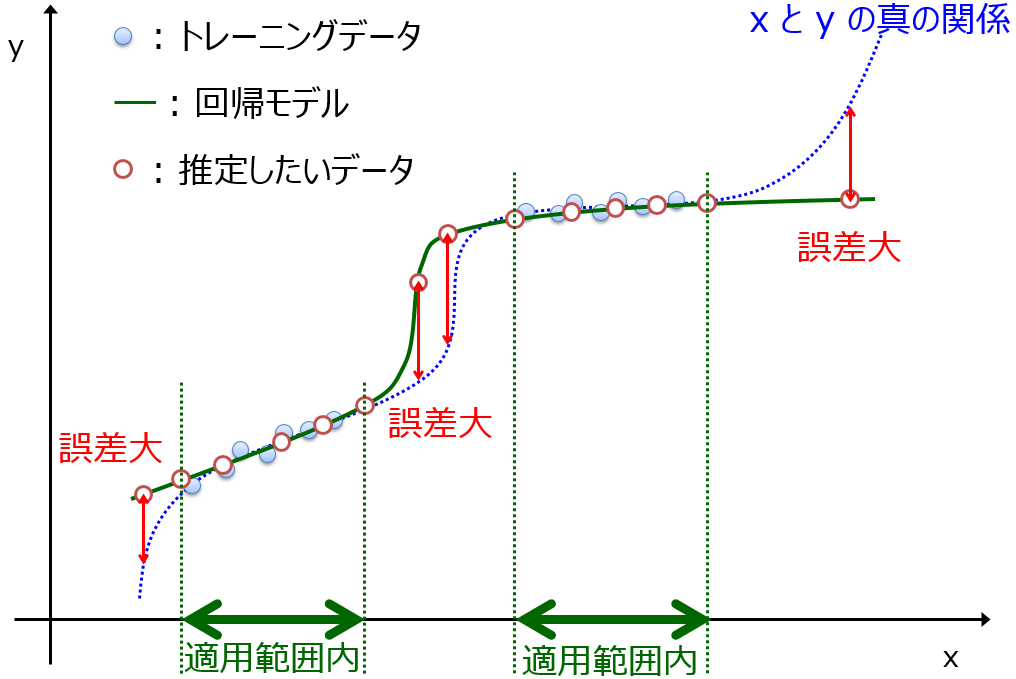
は忘れずに設定しましょう。AD 内の化学構造の推定値のみ、解釈のために使用できます。
2 つ目として、基本的には QSAR モデルや QSPR モデルは非線形ですので、側鎖が同じでも、主骨格が違うと、活性や物性に対して異なる影響になる可能性があります。注意しましょう。
3. 主骨格とか側鎖はどうやって準備すればよいの? → BRICS アルゴリズムで作成できます!
主骨格が決まっていたり、側鎖のフラグメントライブラリをお持ちの方は問題ないですが、お持ちでない方は、BRICS (Breaking of Retrosynthetically Interesting Chemical Substructures) アルゴリズムで作成できます。こちらのプログラミング課題における 20 で扱っていまして、

BRICSアルゴリズムで化学構造をフラグメントに分解できます。これを利用して主骨格や側鎖を作成するとよいでしょう。
こちら https://github.com/hkaneko1985/structure_generator_based_on_r_group に BRICS アルゴリズムでフラグメントを生成するプログラムを追加しました。自由結合手の数の指定もできます。例として、水溶解度 (logS) の測定された化合物群から生成しているプログラムがあります。
必要な方はぜひお使いください。
以上です。
質問やコメントなどありましたら、twitter, facebook, メールなどでご連絡いただけるとうれしいです。


