「DCE tool」に機能を追加しましたので報告します!
追加した機能は、
- 逆解析のための予測用サンプルの生成
- 化学構造モード
です。順に説明します。なお新しい DCE tool はこちら↓からダウンロードをお願いします。
まず DCE tool の基本的な使い方と、最初の機能追加に関してはこちらをご覧ください。
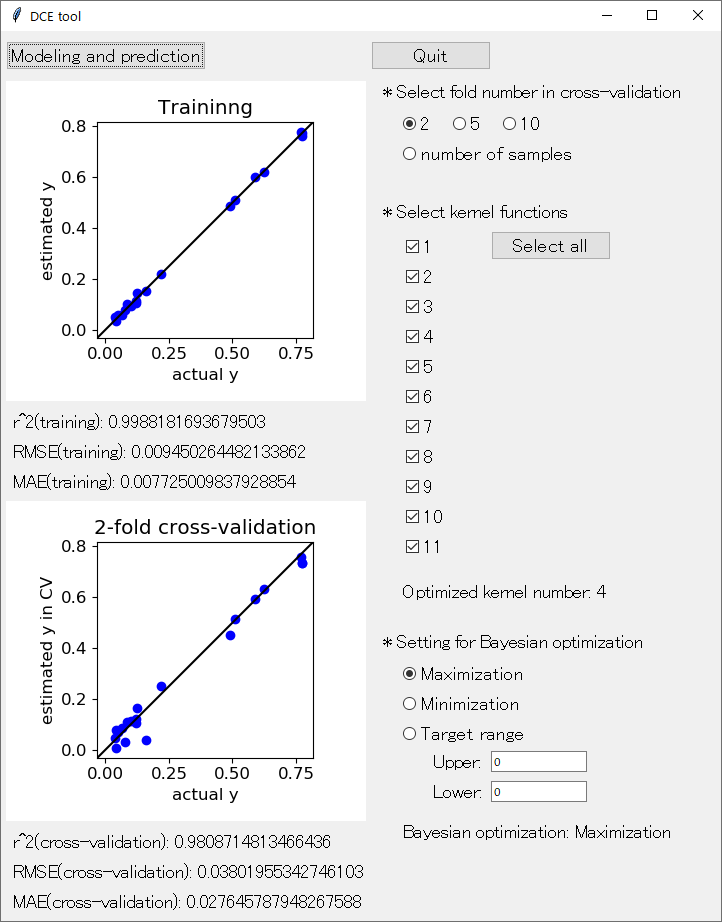
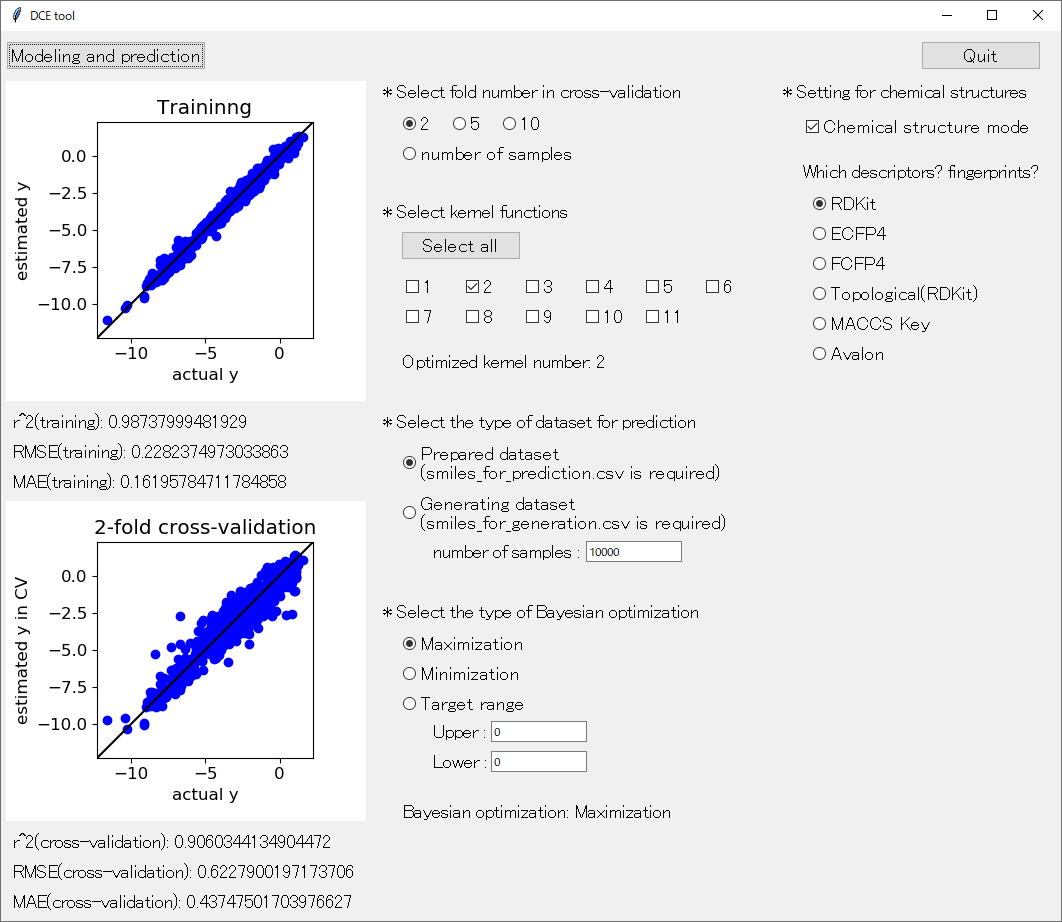
アップデートした DCE tool の起動画面はこんな感じです。
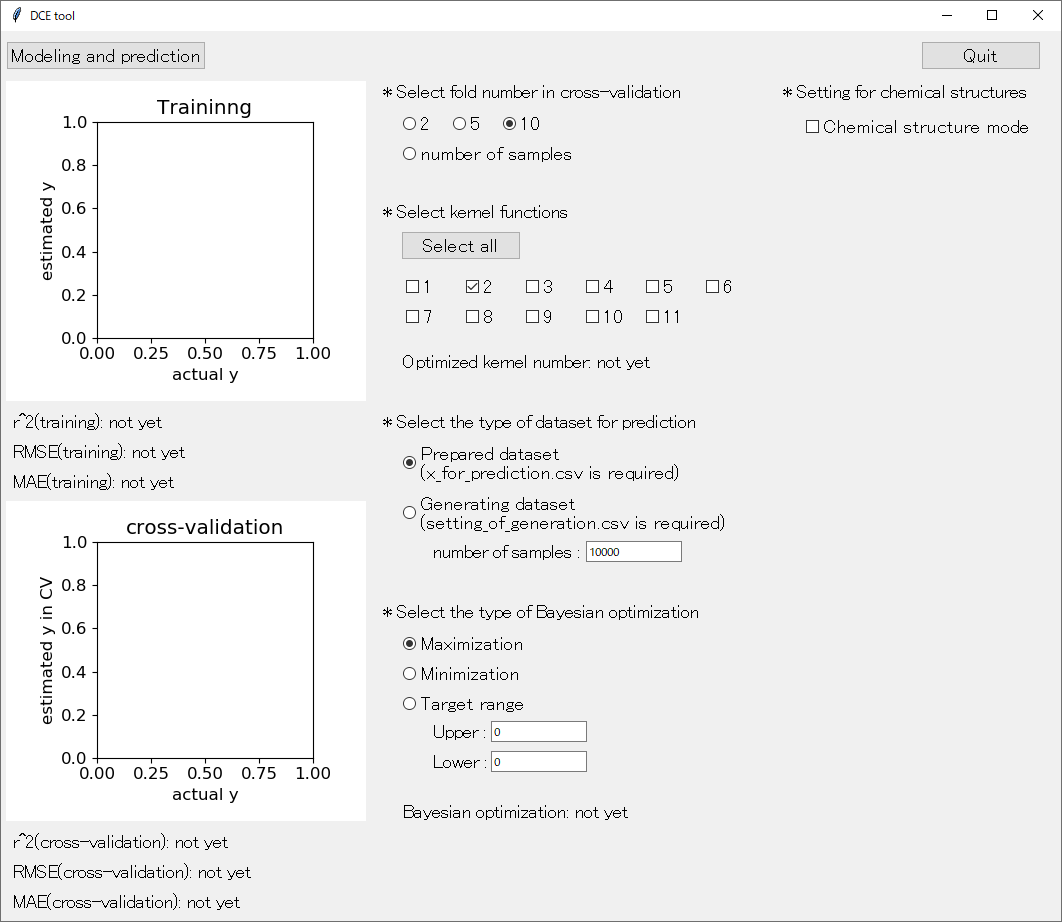
まず、逆解析のための予測用サンプルの生成 「* Select the type of dataset for prediction」 についてです。初期設定では 「Prepared dataset (x_for_prediction.csv is required)」 となっており、これまでと同様に、事前に準備した x_for_prediction.csv のサンプルにおける説明変数 X の値を、ガウス過程回帰 (Gaussian Process Regression, GPR) モデルに入力して、目的変数 Y の値およびその標準偏差を予測します。
一方で、「Generating dataset (setting_of_generation.csv is required)」をチェックすると、x_for_prediction.csv に代わるサンプルが、乱数に基づいて自動的に生成されます。その下の 「number of samples :」 に生成したいサンプルの数を入力してください。サンプル生成のためには 「setting_of_generation.csv」 という csv ファイルが必要です。このファイルは以下のような内容になっています。
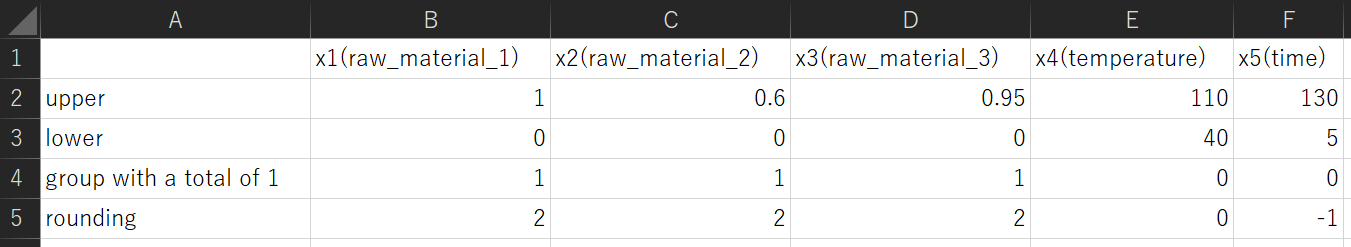
1行目のB列以降である X の名前の部分は、training_data.csv における X の名前とまったく同じにしてください (setting_of_generation.csv には Y はありません)。2 行目の upper には X ごとの上限値を、3 行目の lower には X ごとの下限値を設定してください。たとえば図の x4(temperature) では上限値が 110、下限値が 40 ですので、40 から 110 の間で値が生成されることになります。
4 行目の group with a total of 1 には、特徴量の合計が 1 になってほしい特徴量のグループに、1 から順に同じ番号を入れます。特に関係ない特徴量は 0 としてください。上の図においては、x1(raw_material_1), x2(raw_material_2), x3(raw_material_3) の 3 つの特徴量の和が 1 になるようにサンプルが生成されます。なお、他にも合計を 1 にしたい特徴量の組み合わせがあれば、それらの特徴量は 2 としてください。他にも、3, 4, … と、任意の数のグループを作ることができます。
なお、特徴量の合計を調整する過程で、上限値を超えたり下限値を下回ったりしたサンプルは削除されるため、「number of samples :」 で設定した数より小さくなる可能性があります。
有効数字を設定したい場合は 4 行目の rounding で設定します。不要な場合は、3 行目まででOK です。4 行目ごと (rounding も含めて) 削除してください。有効数字の桁数の指定の仕方として、小数点 m 桁目まで残したい ((m + 1) 桁目を四捨五入したい) 場合は m、10 の n 乗の位まで残したい (10 の (n − 1) 乗の位で四捨五入したい) 場合は -n とします。たとえば上図の例では、x1(raw_material_1), x2(raw_material_2), x3(raw_material_3) は小数点 2 桁目まで残し、x4(temperature) は 10 の 0 乗の位 (一の位) まで残し、x5(time) は 10 の 1 乗の位まで残します。
【Modeling and prediction】 をクリックして実行すると、生成されたサンプルが results フォルダに x_for_prediction_generated.csv という csv ファイルとして保存されます。
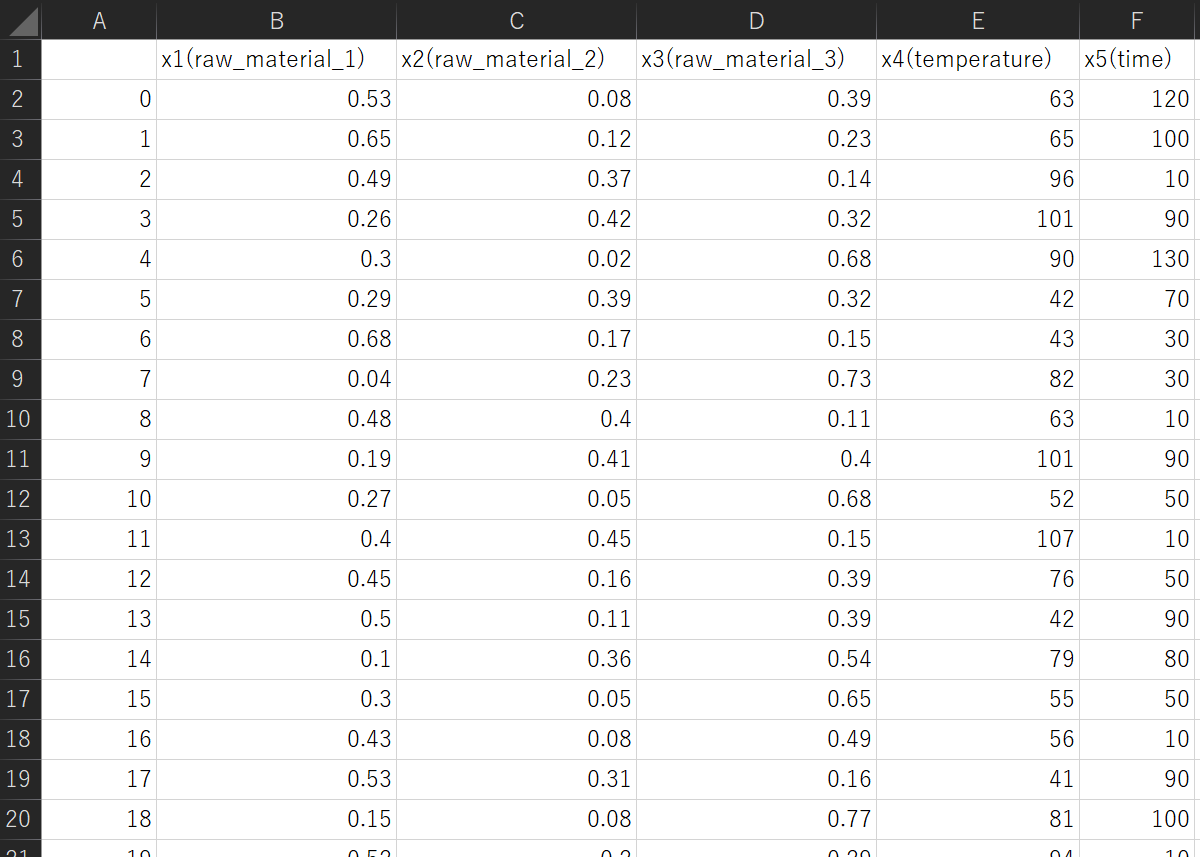
上図のように、特徴量ごとに下限値から上限値までの間に入っていることや、x1(raw_material_1), x2(raw_material_2), x3(raw_material_3) の和が 1 になっていることや、特徴量ごとに有効数字が設定されていることが確認できます。これらの X の値をモデルに入力して Y の値が予測されます。モデル構築・クロスバリデーション・予測やそれらの結果の保存に関しては、これまでと同じです。
続いて、化学構造モード 「* Setting for chemical structures」 についてです。化学構造モードにするためには、その下の 「Chemical structure mode」 をチェックしてください。下図のような画面になります。
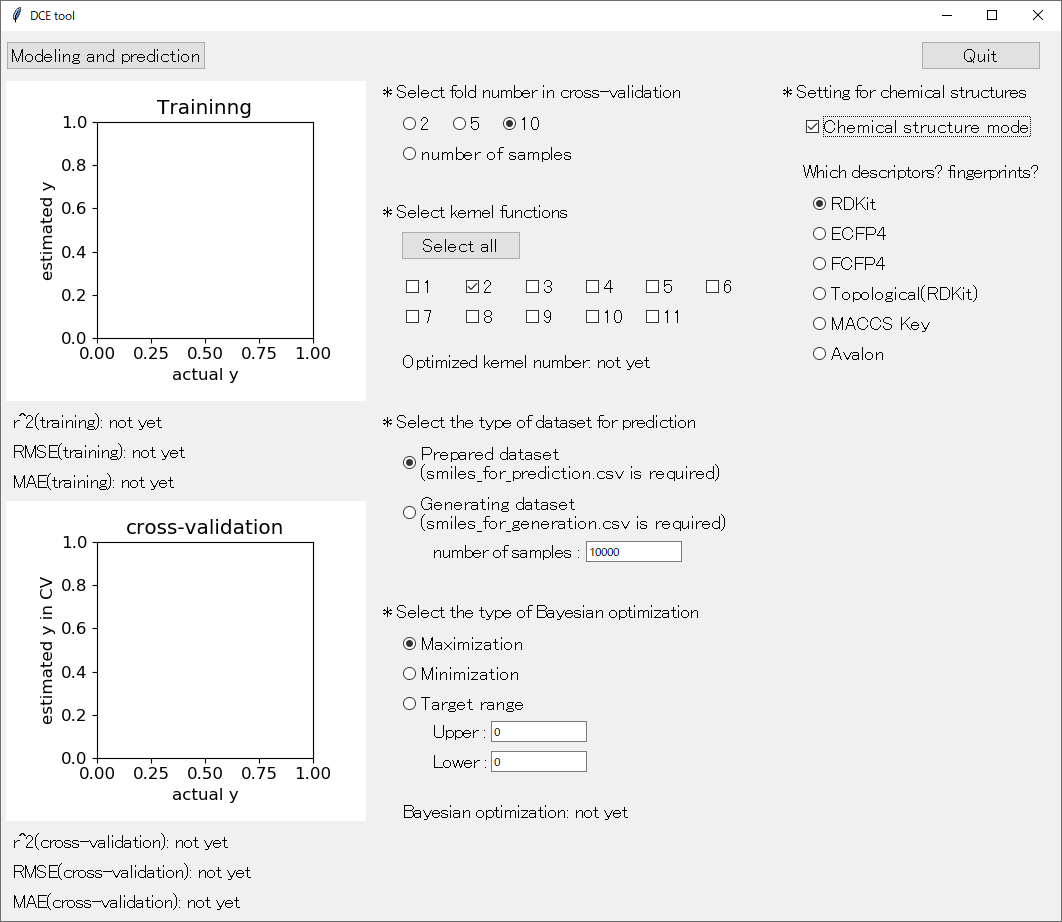
このモードでは、入力するデータが分子になります。分子の化学構造は SMILES で与えてください。
training_data.csv, x_for_prediction.csv の代わりに、それぞれ training_data_smiles.csv, smiles_for_prediction.csv をご準備ください。
- training_data.csv → training_data_smiles.csv: トレーニングデータ。このデータを用いて GPR モデルを構築します
- x_for_prediction.csv → smiles_for_prediction.csv: 予測用のデータ。構築された GPR モデルに入力して、Y の値およびその標準偏差を予測します
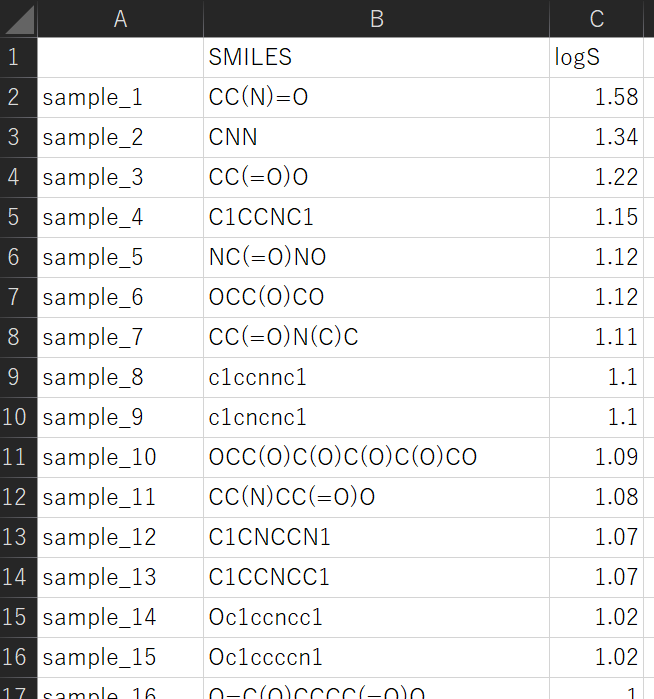
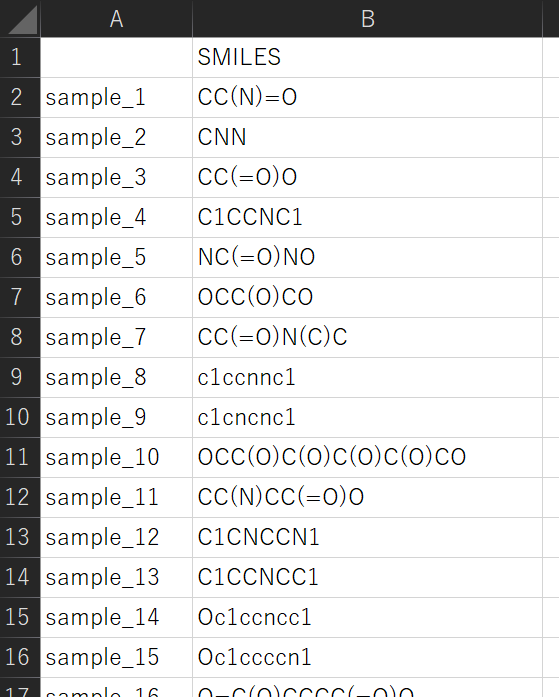
サンプルデータセットとして、以下の図のような training_data_smiles.csv と smiles_for_prediction.csv があります。
training_data_smiles.csv
↓
smiles_for_prediction.csv
↓
これらは T. J. Hou らの論文 (J. Chem. Inf. Comput. Sci., 44, 266–275, 2004) にある 1290 の化合物に対する水溶解度の log 値のデータセットです)。
training_data_smiles.csv は、一番左の列がサンプル名、次の列が SMILES、その次が Y です。一番上の行が変数名で、その下からが実際のデータセットです。smiles_for_prediction.csv は、一番左の列がサンプル名、次の列が SMILES です (Y はありません)。一番上の行が変数名で、その下からが実際のデータセットです。
SMILES から分子記述子を計算します。記述子の種類を、起動画面の右の方にある RDKit, ECFP4, FCFP4, Topological(RDKit), MACCS Key, Avalon の中から一つ選びます。RDKit 以外は fingerprint です。
他の設定をして 【Modeling and prediction】 をクリックして実行すると、まず training_data_smiles.csv, smiles_for_prediction.csv それぞれの SMILES から記述子が計算され、その結果が results フォルダにそれぞれ training_data_descriptor.csv, x_for_prediction_descriptor.csv として保存されます。その後は、これまでの機能と同様にして、記述子 X と Y の間でモデル構築やクロスバリデーションが行われたり、予測用サンプルの Y の値が予測されたりして、それらの結果が results フォルダに保存されます。
最後に、予測用サンプルの生成と化学構造モードを組み合わせることができます。つまり、化学構造生成です。DCE tool では、BRICS (Breaking of Retrosynthetically Interesting Chemical Substructures) アルゴリズムで化学構造が生成されます。
化学構造生成をするためには、化学構造モードにおいて 「* Select the type of dataset for prediction」 の 「Generating dataset (smiles_for_generation.csv is required)」 をチェックしてください。そしてその下の 「number of samples :」 に生成したい化学構造の数を入力してください。化学構造を生成するためには、種になる化学構造の SMILES を 「smiles_for_generation.csv」 という csv ファイルとして準備する必要があります。下図のような csv ファイルを準備しましょう。
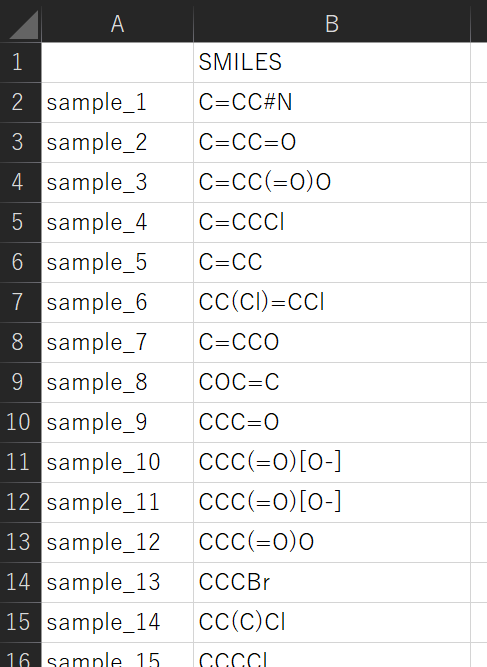
他の設定をして 【Modeling and prediction】 をクリックして実行すると、生成された化学構造が generated_structures.csv として保存されます。ただし、化学構造生成した後に重複した構造を削除するため、「number of samples :」 で入力した数より生成した数は小さい可能性があります。その後の解析や結果の保存については、これまでと同様です。
たとえば次に合成する化学構造の候補として、probabilities が最大となる候補を選択するとよいでしょう。
ぜひ新しい機能が追加された DCE tool をご活用ください。
以上です。
質問やコメントなどありましたら、twitter, facebook, メールなどでご連絡いただけるとうれしいです。