回帰モデルやクラス分類モデルを構築した後は、モデルの逆解析をします。

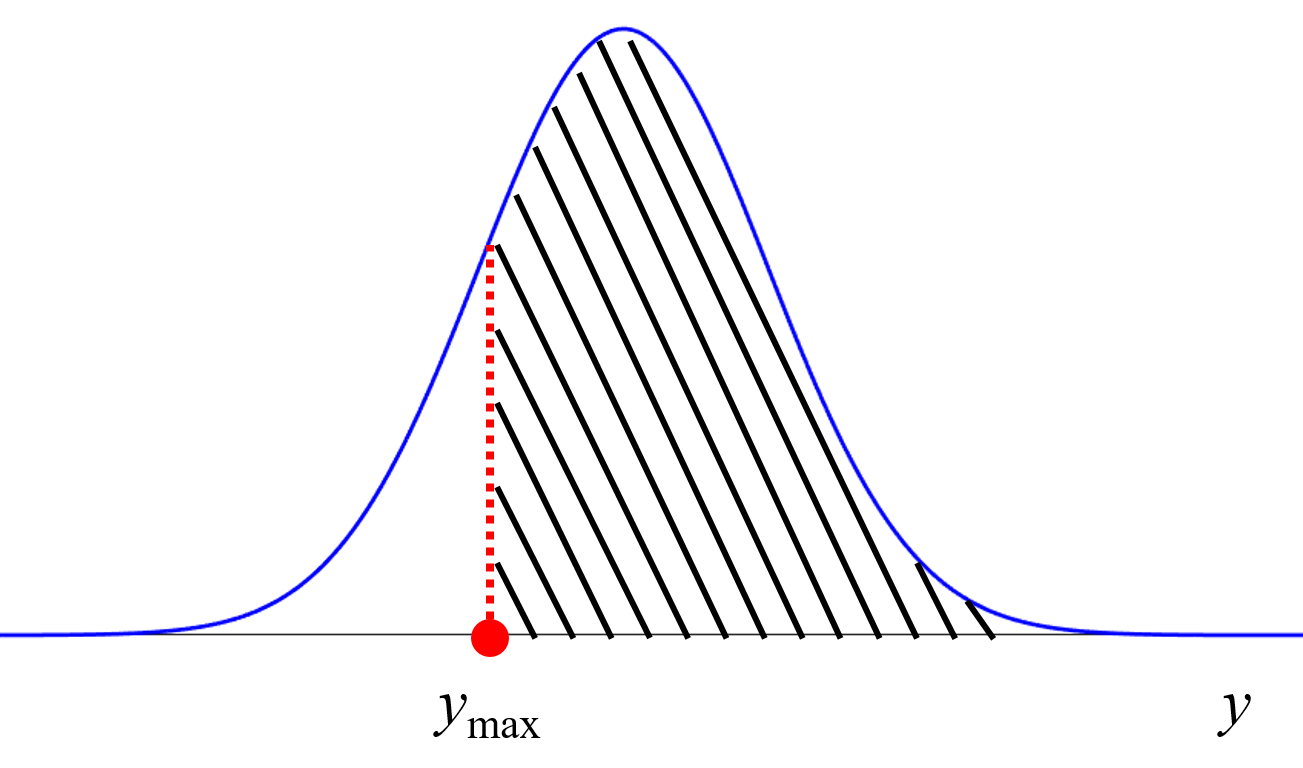
説明変数 (特徴量・記述子など) X のサンプルをたくさん生成して、それらをモデルに入力することで、目的変数 (活性・物性など) Y の値を推定します。推定された値が、よりよい (目標に近い) ようなサンプルを選択します。もちろん、ベイズ最適化のように推定値だけでなく推定値のばらつきを考慮して、目標の範囲内に入る確率をはじめとした獲得関数を計算する方法もあります
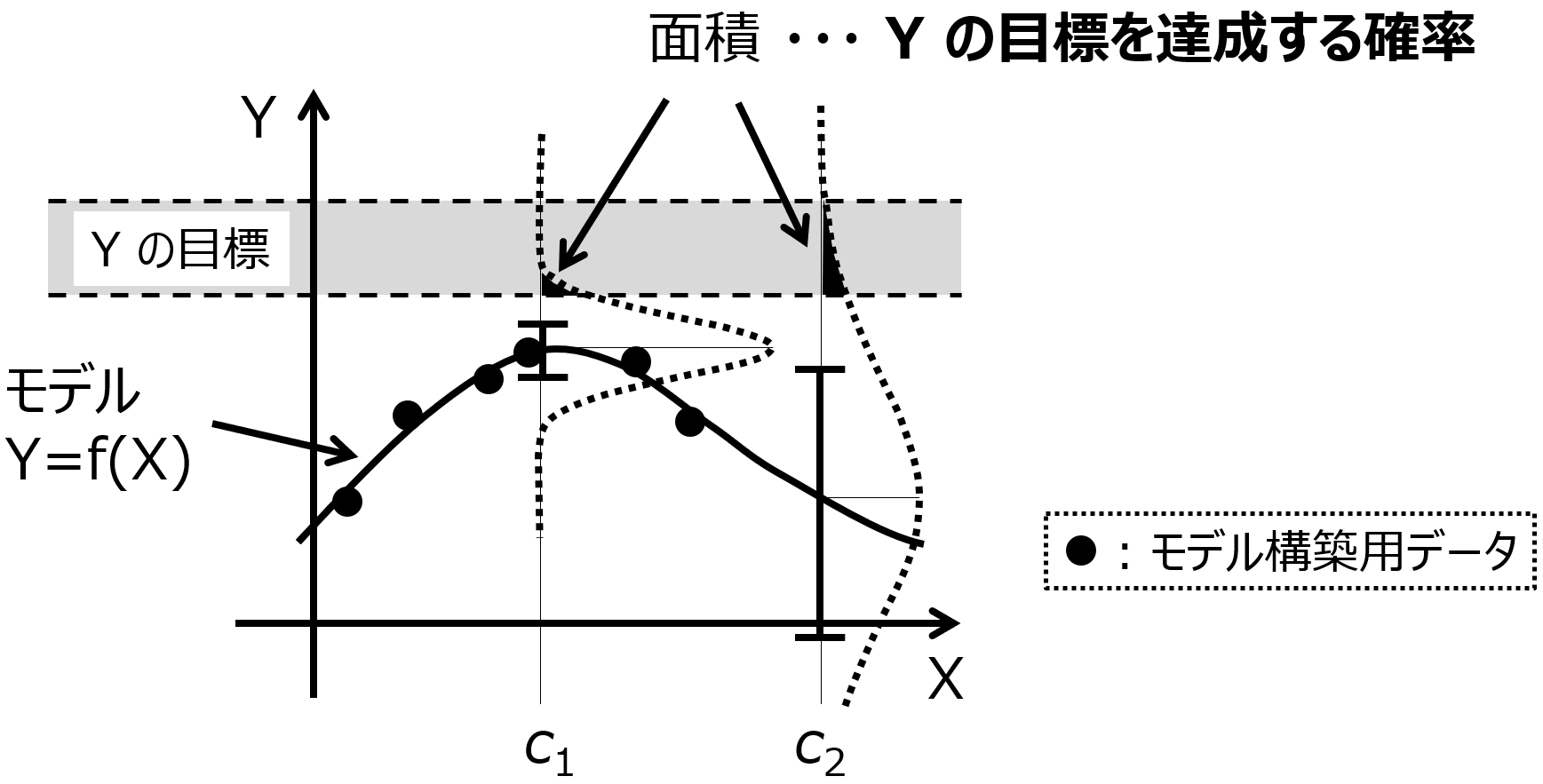
一般的なモデルの逆解析とベイズ最適化の使い分けについてはこちらをご覧ください。

ただいずれにせよ、モデルの解析を行うときには、サンプルをたくさん生成することになります。基本的には、乱数を使ってサンプルを生成します。X のそれぞれに上限と下限を設定して (たとえば既存のサンプルの最大値や最小値など)、上限と下限の間に一様乱数を生成する方法があります。X の間に制限、たとえば組成比のように、いくつかの X を足し合わせて 1 になるといったものがあるときは、生成したあとにサンプル候補を調整します。
生成したサンプルをモデルに入力する前に、モデルの適用範囲 (Applicability Domain, AD) を設定する必要があります。
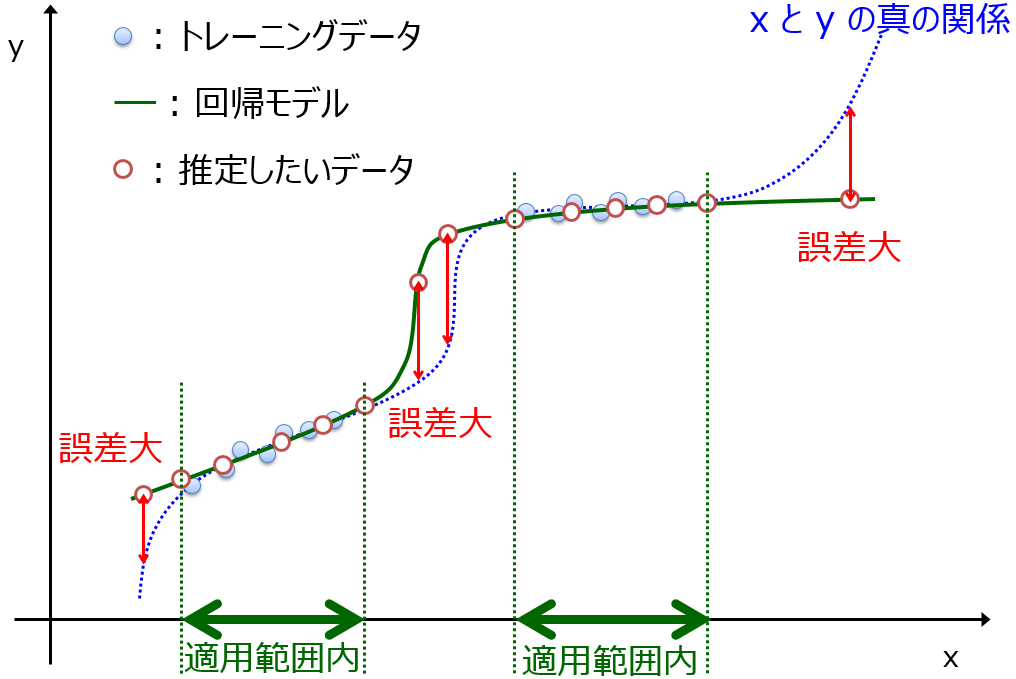
AD 内のサンプル候補のみ、Y の値を予測することになるわけです。AD を考慮すると、一様乱数でたくさんサンプルを生成したときに、もちろん AD 内のサンプルも生成されますが、AD 外のサンプルもたくさん生成されてしまいます。サンプルの生成としては非効率です。
AD は既存のサンプルにもとづいて設定することから、既存のサンプルに近いようなサンプルを生成できれば、 AD 内のサンプルのみが生成されて、効率的といえます。
そこで、既存のサンプルのデータ分布を求めて、その分布に従うようにして、新たなサンプルを生成することを考えます。データ分布を求めるときには、Gaussian Mixture Models (GMM) を活用します。これはデータ分布を、正規分布を重ね合わせたものと仮定して、その正規分布や各正規分布の重みを最適化する方法です。詳細はこちらをご覧ください。
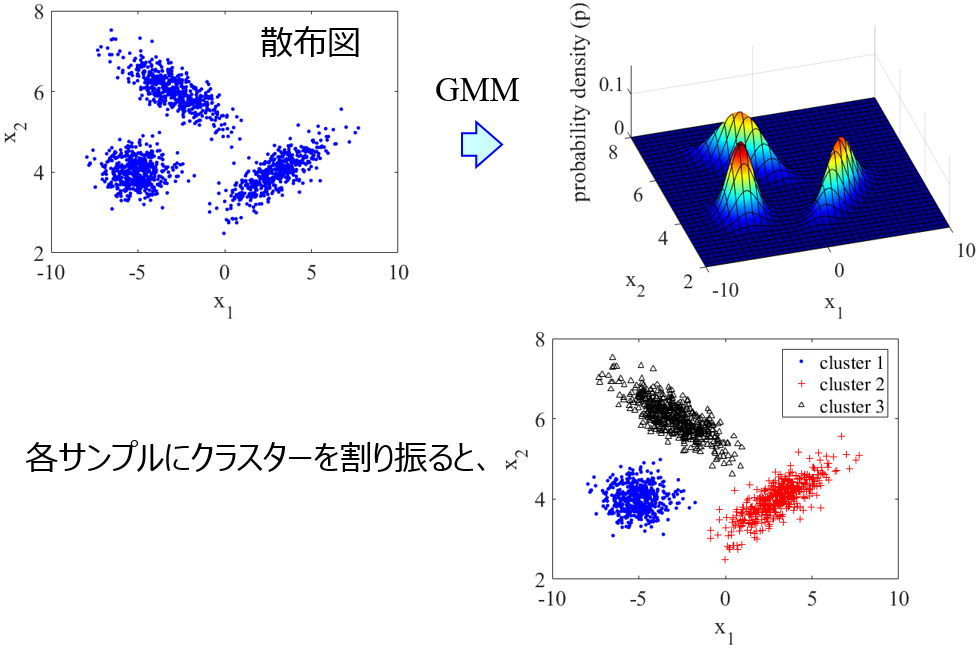
GMM によって正規分布の重ね合わせとしてのデータ分布を計算したあとに、その分布に従うようにサンプルを生成します。
具体的には、GMM に基づいて新たなサンプルを生成するデモンストレーションの Python コード
demo_sample_generation_based_on_gmm.py
を準備しました。以下の URL から、ぜひご利用ください。
デモンストレーションでは、あやめのデータセットで GMM によって正規分布の重ね合わせとしのデータ分布を求めてから、GMM に基づいて新たなサンプルを生成します。たとえば 1 万個のサンプルを生成すると、以下の図のような分布になります。
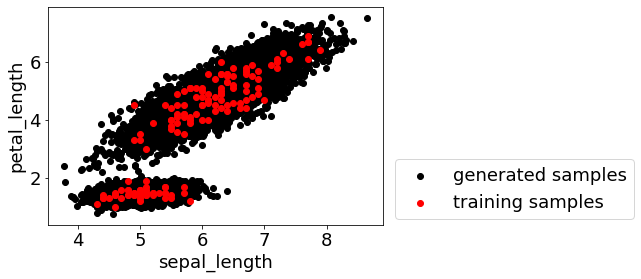
プログラム demo_sample_generation_based_on_gmm.py において、
file_name = ‘iris_without_species.csv’
にて既存のデータベースの csv ファイルの名前を指定し、
number_of_samples_generated = 10000
にて生成するサンプル数を指定すれば、あやめのデータセット以外でも活用できます。csv ファイルは iris_without_species.csv と同様の形式にしてください。
参考になりましたら、ぜひご活用いただければと思います。
以上です。
質問やコメントなどありましたら、twitter, facebook, メールなどでご連絡いただけるとうれしいです。
