Structure Generator based on R-Group (SGRG) という化学構造を生成する Python プログラムへの、新たな機能追加です。今回の機能追加でバージョンを 1.0 → 2.0 としました。
SGRG に関するこれまでの流れはこちらをご覧ください。


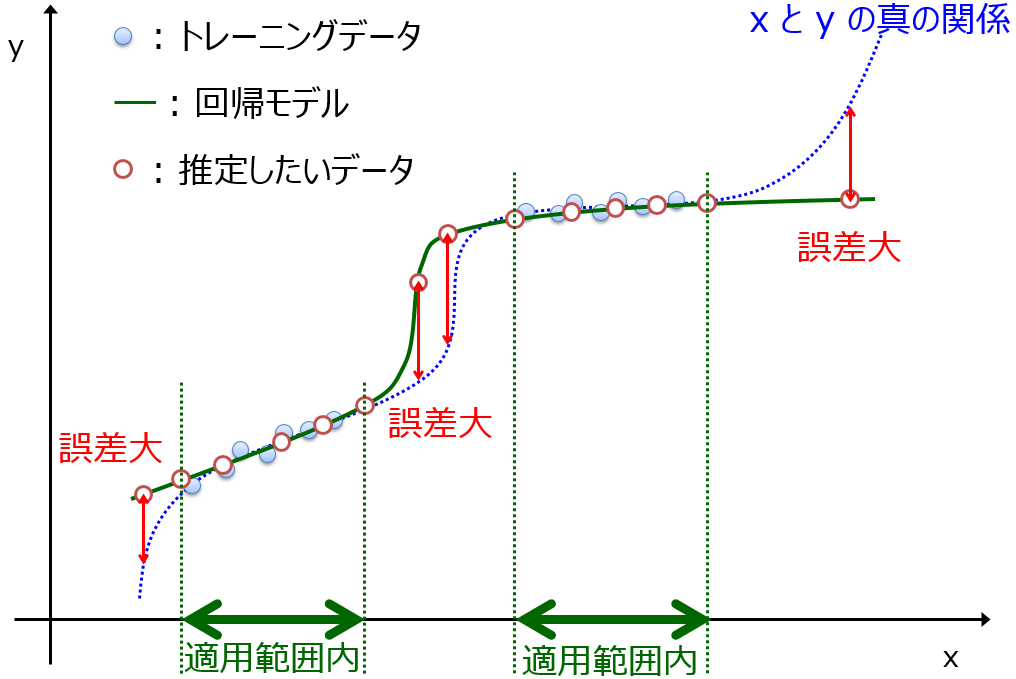
前回は、回帰モデルから計算された物性や活性の推定値が大きくなるように、遺伝的アルゴリズム (Genetic Algorithm, GA) によりメインの骨格と側鎖を最適化するプログラム structure_generator_based_on_r_group_ga.py では、確かに物性・活性の推定値が向上する化学構造が得られるのですが、モデルの適用範囲 (Applicability Domain, AD) は考慮されていませんでした。
そこで今回は、AD ももちろん考慮されている、ベイズ最適化 (Bayesian Optimization, BO)
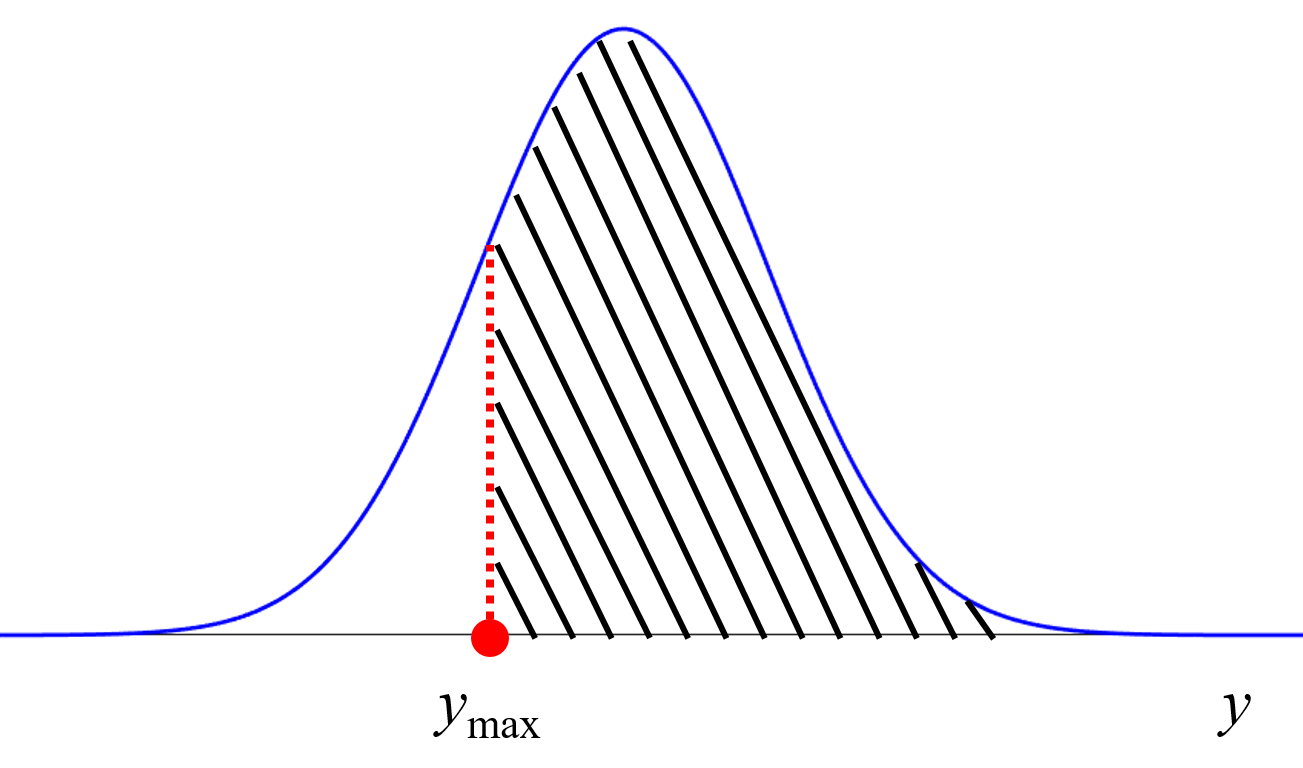
により化学構造を最適化する機能を追加しました。ガウス過程による回帰 (Gaussian Process Regression, GPR)
をして、事前に決めた物性・活性の目標範囲に入る確率を、遺伝的アルゴリズム (Genetic Algorithm, GA) の目的関数にして、構造を最適化するわけです。結果的に、物性・活性の目標を満たす確率の高い化学構造が得られます。
こちらからご利用ください。
structure_generator_based_on_r_group_ga.py において
method_name = ‘gp’
とすると、今回の機能を利用できます。その下の
target_range = [1, 3]
で目標の範囲を設定してください (上の例では、目標範囲は 1 から 3 までになります)。他のやり方は前回

と同じです。とりあえず実行してどんな構造が生成されるか、確認するとよいと思います。
さらに今回は物性・活性が複数の場合にも、ベイズ最適化によりすべての物性・活性の目標を同時に満たす確率の高くなるような化学構造を生成するプログラムも開発しました。structure_generator_based_on_r_group_ga_multi_y.py です。目的変数ごとの目標範囲を設定するため、settings_of_target_ys.csv という csv ファイルで目標範囲を設定し、同じフォルダ (ディレクトリ) に置く必要があります。最初の settings_of_target_ys.csv では目的変数が 3 つあるときに、
- 1 つ目: 目標範囲 1 から 5 まで
- 2 つ目: 目標範囲 20 から 40 まで
- 3 つ目: 目標範囲 -40 から -20 まで
となっています。これらの値を変更したり、目的変数が 4 つ以上の場合は、新たに列を作成したりしてください。他のやり方は前回

と同じです。こちらも、とりあえず実行してどんな構造が生成されるか、確認するとよいと思います。サンプルデータセットとして、水溶解度が測定された化合物において、仮想的な物性を 2 つ追加して目的変数の数を 3 つにした molecules_with_multi_y.csv があります。
ぜひご活用ください。
以上です。
質問やコメントなどありましたら、twitter, facebook, メールなどでご連絡いただけるとうれしいです。


