以前に、Structure Generator based on R-Group (SGRG) という化学構造を生成する Python プログラムを公開しました。
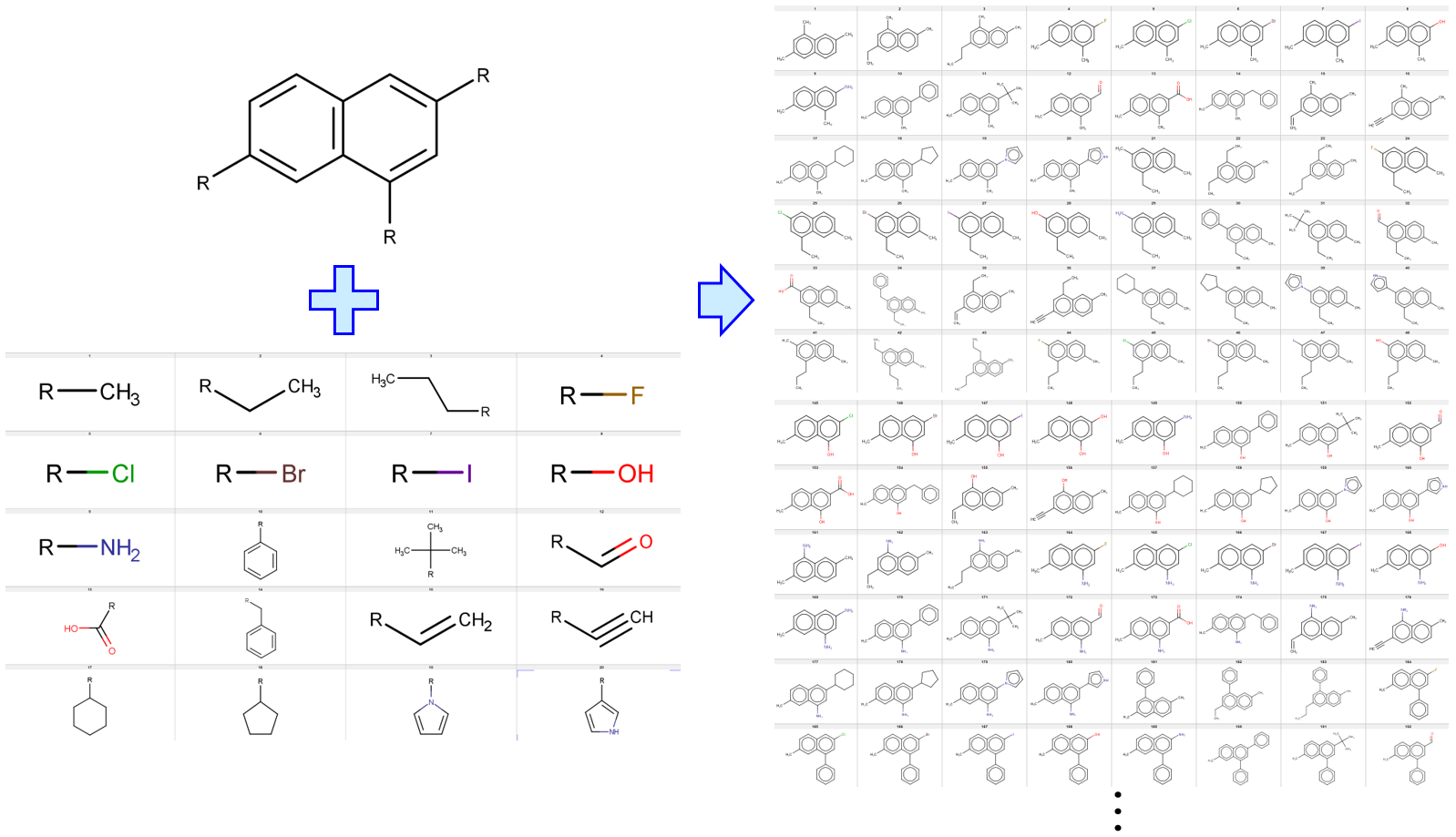
メインの骨格を一つに設定して、その自由結合手に結合する側鎖を、フラグメントの候補の全ての組み合わせとして、化学構造を生成するプログラムでした。
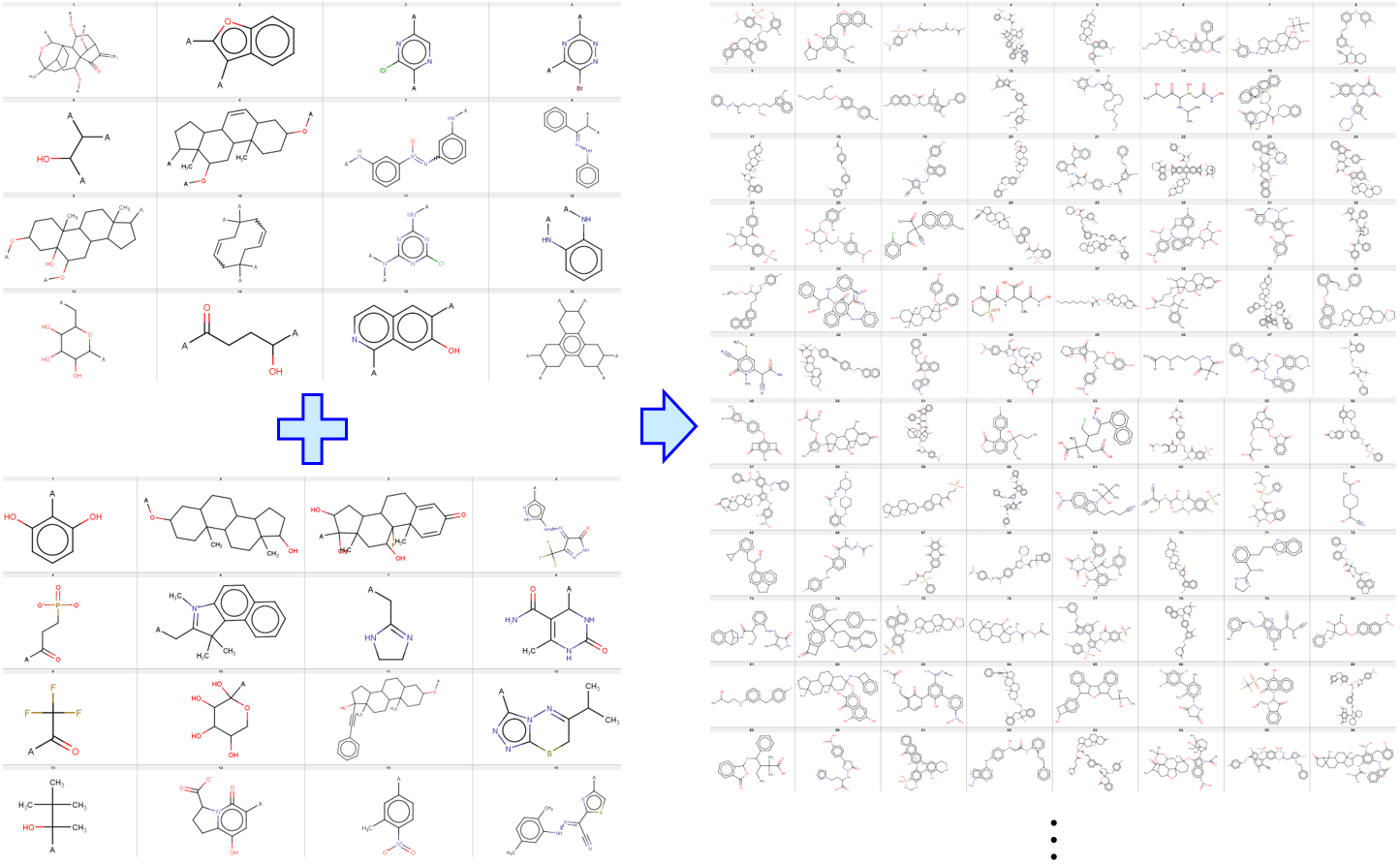
その後に、メインの骨格がいくつかあり、フラグメントの候補もあるときに、それらをランダムに選びながら化学構造を生成する Python プログラムを公開しました。
Google の collaboratory でも実行できるようにしました。

ランダムにメインの骨格が選ばれると、自由結合手の数が決まり、その数だけランダムにフラグメントを選び構造を作ります。これを繰り返します。
今回は、回帰モデルから計算された物性や活性の推定値が大きくなるように、遺伝的アルゴリズム (Genetic Algorithm, GA) によりメインの骨格と側鎖を最適化するプログラムです。
Python プログラム structure_generator_based_on_r_group_ga.py はこちらの Github にありますので、とりあえず実行してどんな構造が生成されるか、確認するとよいと思います。なお structure_generator.py も一緒のフォルダ (ディレクトリ) に置いてください。サンプルデータセットとして、水溶解度が測定された化合物である molecules_with_logS.csv があります。
回帰モデルを構築するための化合物データセットも、メインの骨格の候補も、側鎖のフラグメントの候補も、SMILES で読み込む必要があります。SMILES についてはこちらをご覧ください。
回帰モデルを構築するための化合物データセットの形式は、サンプルデータセットの molecules_with_logS.csv をご覧ください。同様の形式のデータセットを準備すれば、同じようにプログラムを利用できます。今回のプログラムは物性や活性の値を大きくするように化学構造を生成するプログラムなので、もし物性や活性の値を小さくするように化学構造を生成したいときは、あらかじめデータセットにおける物性や活性に -1 をかけておくとよいでしょう。
回帰分析手法として PLS と SVR がありますが、
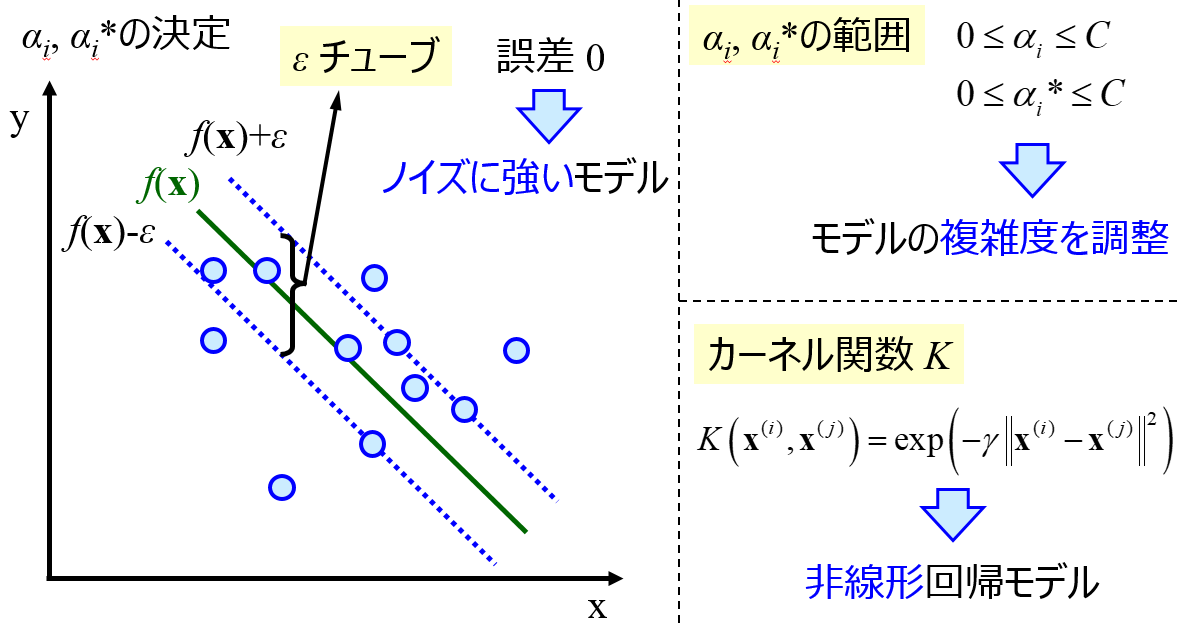
他にも追加することができます。
サンプルのメインの骨格の候補として sample_main_fragments.smi、サンプルの側鎖のフラグメントの候補として sample_sub_fragments.smi がプログラムと一緒にありますので、ぜひご利用ください。
回帰モデルを構築するための化合物データセットのファイルと、sample_main_fragments.smi と、sample_sub_fragments.smi を、プログラム structure_generator_based_on_r_group_ga.py と同じフォルダ (ディレクトリ) におき、プログラムを実行してください。
プログラム structure_generator_based_on_r_group_ga.py では、number_of_iteration_of_ga で GA による構造生成を何回繰り返すかを指定できます。GA の個体数である number_of_population × number_of_iteration_of_ga の数だけ化学構造が生成されるわけです。
生成された化学構造は SMILES で、回帰モデルによる推定値と一緒に generated_molecules.csv という名前の csv ファイルに保存されます。
メインの骨格の候補や側鎖のフラグメントの候補をご自身で準備して、ファイルの名前をそれぞれ sample_main_fragments.smi, sample_sub_fragments.smi とすれば、それらのメインの骨格や側鎖のフラグメントで、化学構造を生成できます。
ぜひご活用ください。
以上です。
質問やコメントなどありましたら、twitter, facebook, メールなどでご連絡いただけるとうれしいです。


