(アイキャッチ画像は、Midjourney にて chemical structures, molecules, organic chemistry, benzene, carbon, hydrogen で描画)
データ解析・機械学習に基づいた分子設計をしているとき、分子記述子 x と物性・活性 y との間で回帰モデル y=f(x) を構築したときなど、新たな化学構造をサクッと生成したいときがあると思います。ここでは、化学構造の表記方法の一つである SELFIES に基づいて、シンプルに新規化学構造を生成する Python コードを作成しましたので、DCEKit で共有します。

SELFIES は SMILES のように化学構造を記述する方法の一つです。元論文はこちらであり、日本語でも SELFIES の分かりやすい解説記事があります。
Github はこちらであり、conda-forge から SELFIES のパッケージをインストールできます。
SELFIESの大きな特徴の一つとして、元論文の一部を引用すると、
The major challenge here is that the standard strings molecular representation SMILES shows substantial weaknesses in that task because large fractions of strings do not correspond to valid molecules. Here, we solve this problem at a fundamental level and introduce SELFIES (SELF-referencing Embedded Strings), a string-based representation of molecules which is 100% robust. Every SELFIES string corresponds to a valid molecule, and SELFIES can represent every molecule.
とあり、多少文法が異なっていても、最低限のルールを守っていれば化学構造に変換できる、というのがあります。例えば SMILES 記法では、適当な文字列にしただけなど、SMILES の文法から少しでも外れると、化学構造に変換できません。一方で、SELFIES 記法であれば、多少適当に、例えばランダムに文字列 [*] を並べても、化学構造に (適切な SMILES に) 変換できます。
この SELFIES の特徴を生かして、元の分子の化学構造に基づきながらも、適当に化学構造を生成する Python コードを作成しました。DCEKit の
demo_structure_generation_on_selfies.py
です。こちらからダウンロードできます。
ここでは、元になる分子の化学構造 (SMILES) を用いて、それぞれの化学構造を SELFIES で表現し、それらの構造で使われている SELFIES の文字列の単語 [*] を取得します。取得した [*] をランダムに生成し、得られた SELFIES に基づいて化学構造を生成します。コードでは SMILES に変換して csv ファイル (generated_smiles.csv) として保存しています。
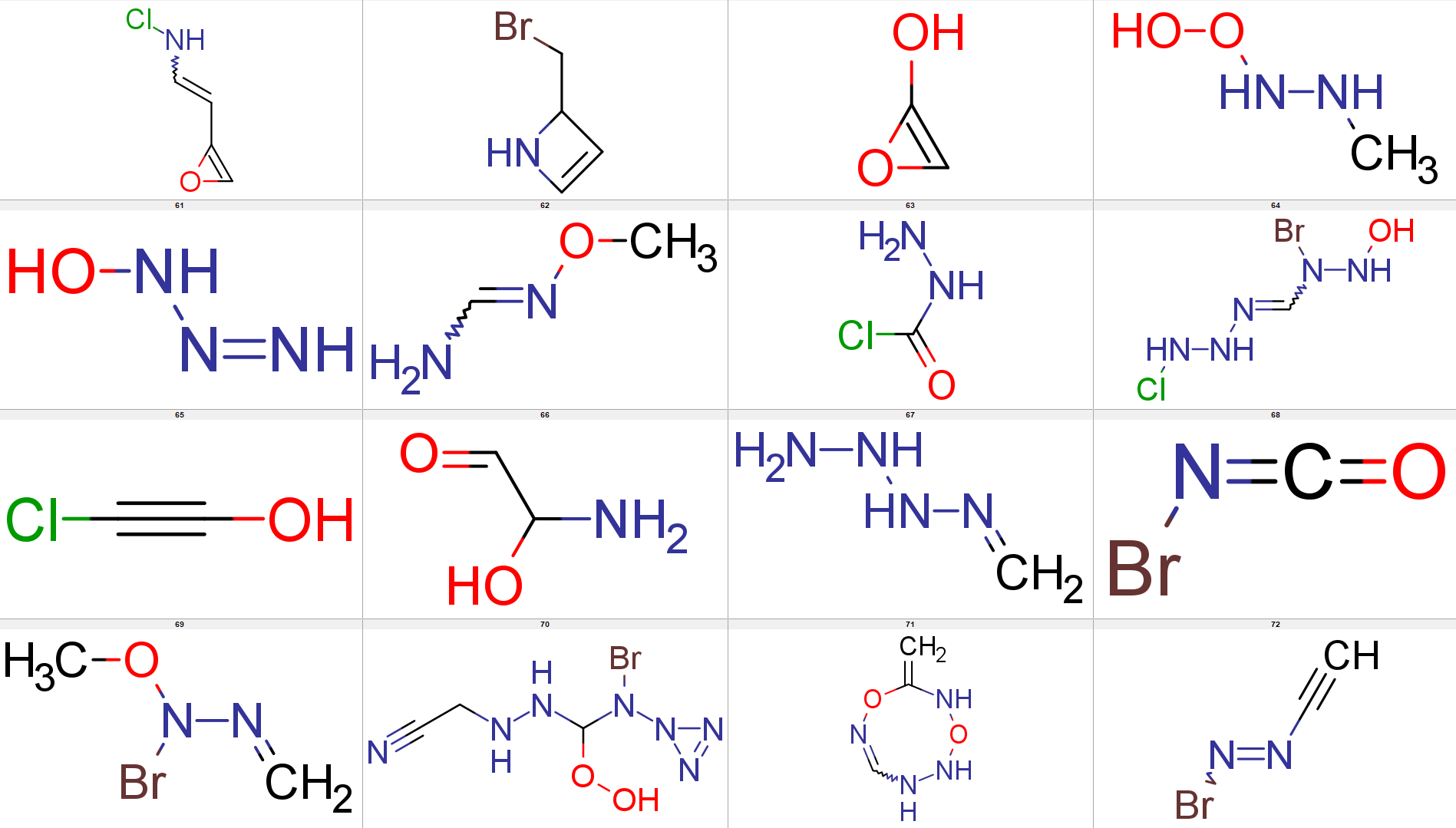
SELFIES を用いる上で注意することがあります。RDKit でも用いられている canonical SMILES の記法では、一つの SMILES に対して唯一の化学構造を定義でき、逆に一つの化学構造に対して唯一の SMILES を定義できます。もちろん、SMILES では配座異性体の違いなどの三次元構造としての違いを区別できないため、二次元で区別できる化学構造にはなりますが、SMILES と化学構造が一対一に対応します。一方で SELFIES 記法では、2つの異なる SELFIES が、(二次元でも) 同じ化学構造になることがあります。
そのため、ランダムに (異なる) SELFIES を生成したとき、もちろん SELFIES であれば化学構造に変換できますが、これまで生成した化学構造と重複することがあります。例えば、SELFIES に基づいて 10,000 構造を生成しても、重複を省くと 3,000 構造しか残らない、といったことが起こります。
今回の Python コードでは、重複を省いた化学構造 (SMILES) を保存するようにしています。
Python コードで使用しているライブラリは、NumPy, pandas, RDKit, SELFIES のみであり、サクッと新たな化学構造を生成できます。ぜひご活用いただけますと幸いです。
以上です。
質問やコメントなどありましたら、twitter, facebook, メールなどでご連絡いただけるとうれしいです。


